Cara Desain Primer menggunakan Primer3Plus, untuk Polymerase Chain Reaction (PCR)
Primer merupakan potongan DNA pendek utas tunggal (oligonukleotida), panjang primer yang umum digunakan hanya berkisar antara 20 sampai 25 basa. Primer merupakan salah satu bahan esensial yang diperlukan dalam Polymerase Chain Reaction (PCR). Primer berfungsi untuk menginisiasi Polymerase Chain Reaction (PCR). Juga berfungsi untuk membatasi daerah yang akan diamplifikasi pada reaksi PCR.
Tanpa primer, reaksi PCR tidak akan terjadi meskipun enzim dan komponen lainnya sudah tersedia. Idealnya primer memiliki urutan basa nukleotida yang tepat berpasangan dengan urutan basa DNA target yang akan diamplifikasi, dan tidak menempel pada bagian lainnya yang bukan target (mispriming). Disamping itu juga harus memperhatikan susunan nukleotidanya, seperti %GC, dan selisih Temperature Mealting antara Primer Forwad dan Primer Reverse yang tidak terlalu jauh.
Banyak perangkat lunak (software) yang dapat kita gunakan untuk mendesain primer untuk PCR, ada Primer3Plus, PerlPrimer, dan masih banyak lagi. Namun kali ini saya akan sharing cara mendesain primer menggunakan Primer3Plus. Kelebihan Primer3Plus diantaranya adalah software ini berbasis online, sehingga tidak perlu diinstal pada komputer, alias hanya membutuhkan koneksi internet. Kelebihan lainnya adalah pada Primer3Plus juga banyak parameter yang dapat dimodifikasi sesuai selera, jika ingin mendesain primer dengan lebih advance lagi.
- Siapkan: Komputer yang memiliki akses internet, memiliki web browser (seperti Google Chrome, Internet Explorer, Mozilla, atau browser apa saja), dan telah terinstal program MEGA,
- Buka http://www.ncbi.nlm.nih.gov melalui internet browser, kemudian cari nama organisme yang akan didesain primernya. Contohnya Acropora tennuis
- Sekuen target yang diperoleh di NCBI, kemudian disimpan dengan cara klik Send, pilih Complete Record, pada Choose Destination pilih File, pada Format pilih FASTA, lalu klik Create File. Maka file fasta akan tersimpan di komputer yang kita gunakan. File yang dihasilkan berekstensi *.fasta, biasanya bernama sequence.fasta
- Contoh Acropora tenuis dengan kode akses NC_0033522.1, dengan terget gen mitokondria ND1
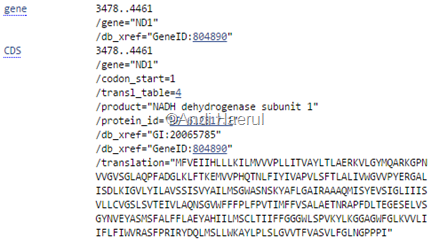
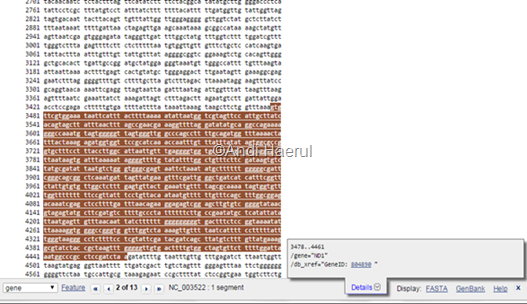
- Masih pada NCBI Schrol ke bawah dan cari posisi gen target, contohnya Gen ND1. Maka akan mengetahui posisi gen berada pada basa keberapa sampai keberapa. Dalam contoh dapat kita lihat bahwa posisi gen ND1 terletak pada basa 3.478 sampai 4.461. Jika gene kita klik maka akan memperlihatkan posisi gen tersebut dalam runutan nukleotidanya (gambar bawah)
- File hasil download tadi kemudian dibuka dengan menggunkan program MEGA, pada program ini kita dapat mengetahui berapa panjang total gen secara keseluruhan (Complete Genome). Pada contoh terlihat bahwa panjang sekuen complete genome Mithocondria karang Acropora tennuis sebesar 18.338 bp.
- Lalu buka alamat website Primer3Plus http://www.bioinformatics.nl/cgi-bin/primer3plus melalui web browser
- Pada kotak Paste source sequence below, copykan sekuen target dari program MEGA. Bisa juga dengan cara menguploadnya dari file fasta yang tersimpan di komputer melalui Choose File lalu Upload File.
- Pilih jenis primer yang kita ingin desain, sense primer (left primer), probe untuk hibridisasi (internal oligo) dan antisense (right primer). Kotak yang ada di bawahnya bisa diisi jika sudah memiliki kandidat primer.
- Tentukan beberapa parameter lain jika diperlukan seperti:
- Excluded Regions (daerah dimana primer tidak diinginkan menempel), cara mengisinya adalah awal nukleotida sekuen yang tidak diinginkan, panjang sekuen yang tidak diinginkan.
- Targets (primer harus mengapit daerah tersebut), cara mengisinya adalah awal nukleotida sekuen target, panjang sekuen target. Pada contoh: 3478, 750, artinya Primer yang kita desain diharapkan untuk mengamplifikasi pada nukleotida ke-3.478 sampai nukleotida ke-4.228 dengan panjang 750 bp.
- Included Region (kedua primer harus berada di dalam daerah ini).
- Beberapa pilihan yang lebih kompleks juga dapat digunakan sesuai kebutuhan, melaui tab General Settings, Advance Settings, Internal Oligo, Penalty Weights dan Sequence Quality.
- Jika semua opsi sudah terisi, tekan tombol Pick Primers untuk memulai pembuatan primer.
- Primer3Plus akan memberikan beberapa alternatif pasangan primer yang dapat kita pilih, biasanya terdiri dari 5 pasang. Akan tetapi biasanya yang paling bagus adalah pasangan pertama.
- Pada gambar di atas terlihat pasangan primer pertama. Memuat Nama Left Primer (Primer Forwad) dan Right Primer (Primer Reverse), sekuen primer, panjang primer (Length), temperature mealting (Tm), % GC, posisi penempelan primer pada sekuen target, Ukuran produk PCR (Product size)
- Gambar di atas memperlihatkan daerah penempelan primer
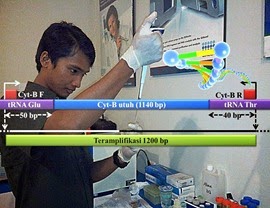
- Contoh di atas memperoleh sepasang primer yakni Primer_F GGAACCTCCGAGACTTTTTGT, dan Primer_R ATTTTACCGGAGAAAGTCACC. Pasangan primer inilah yang digunakan untuk mengamplifikasi sequen target, dengan menggunakan metode Polymerase Chain Reaction (PCR).
- Primer tersebut diperoleh dari proses sinthesis










No comments